Il ruolo dell'anticodone
Nel processo della sintesi proteica nominiamo le molecole principali ma dimentichiamo il ruolo dell’anticodone. Una sequenza di appena tre basi azotate caratteristica per ogni tRNA che trasporta un amminoacido da aggiungere alla catena polipeptidica in formazione. Senza la presenza di queste tre basi non sarebbe possibile legare l’amminoacido e proseguire la traduzione dell’mRNA.
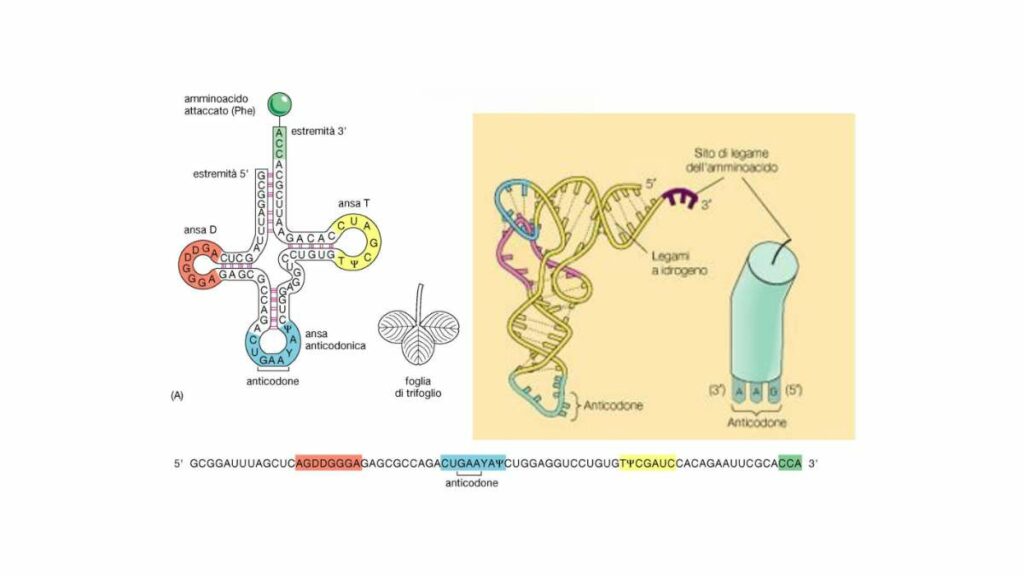
Questa sequenza così cruciale per la sintesi delle proteine ha una precisa collocazione nella struttura dell’RNA di trasporto. Questa tipologia di acido ribonucleico è piccolo e composto da soli 70-90 nucleotidi ma ha una forma caratteristica a trifoglio. Le tre basi sono proprio al centro di una delle tre “foglie”, o meglio anse per usare il termine preciso.
L’anticodone nella struttura del tRNA
Questo particolare RNA è a singolo filamento, ma grazie alla presenza nella sua sequenza di regioni che si autocompletano acquista una struttura tridimensionale. Si formano infatti dei brevi tratti dove il filamento si ripiega formando tre anse allungate. A tenere assieme queste regioni a doppio filamento sono dei legami idrogeno fra le basi complementari.
All’estremità 3′ tutti i tRNA presentano la sequenza CCA-3′, che è dove si lega all’amminoacido da portare ai ribosomi. Questa parte della molecola si chiama infatti stelo accettore. Esaminando le zone a doppio filamento troviamo in posizione centrale tra le tre l’ansa dell’anticodone, chiamato così perché complementare al codone dell’mRNA.
A sinistra di questo loop si trova l’ansa D, che ha il compito di mediare il riconoscimento dell’RNA trasnfer da parte dell’enzima aminoacil-tRNA sintetasi. Si tratta di una regione ricca di diidrouridina, una base derivata da modificazione post-trascrizionali. Dopodiché a destra dell’ansa centrale troviamo altri due loop, uno di lunghezza variabile e uno di dimensioni pari all’ansa D.
L’ansa più grande si chiama anche braccio T e contiene una sequenza molto conservata tra i tRNA di diversi organismi. Il suo ruolo è mediare il l’interazione fra il tRNA e il ribosoma quando arriva sul sito A. Infine L’ultimo loop infine di solito presenta una base chiamata pseudopurina, anch’essa modificata da un enzima.
Il riconoscimento della sequenza complementare
Già a livello della sequenza d’inizio dell’mRNA si rende necessario l’accoppiamento con il rispettivo anticodone. Poiché il primo amminoacido di ogni catena polipeptidica tradotta è la metionina (Met) e la codificano le tre basi AUG, quelle complementari saranno UAC. Una volta che questa tripletta si aggancia all’RNA messaggero sul sito A del ribosoma c’è il vero inizio della traduzione.
Ancora unito al filamento di mRNA il tRNA giunge poi al sito P dove resterà fino all’arrivo del successivo che si lega alla seconda tripletta della sequenza. A questo punto quando anche questo arriva al sito P il primo RNA transfer può passare al sito di uscita, indicato con la lettera E. La catena di amminoacidi si trasferisce sul secondo tRNA.
Poiché come sappiamo il codice genetico è degenerato, c’è più di un anticodone per lo stesso amminoacido. Per esempio per la serina ci sono quattro triplette possibili che possiamo trovare sull’RNA messaggero. Di conseguenza avremo quattro possibili appaiamenti con i tRNA che la portano (UCU-AGA, UCC-AGG, UCA-AGU e UCG-AGC).
In teoria dovrebbe esserci un numero di RNA transfer pari al numero delle triplette possibili, ma controllando si scopre che sono di meno. La spiegazione sta nella cosiddetta ipotesi del vacillamento, proposta da Francis Crick, lo stesso biologo molecolare a cui si deve la scoperta della struttura del DNA.
Un anticodone può legare più di una tripletta
Sappiamo che nelle triplette sono solo i nucleotidi in prima e seconda posizione a determinare l’amminoacido necessario. Infatti quando le mutazioni in terza posizione sono silenti e la proteina sintetizzata risulta funzionale. Inoltre avere il terzo nucleotide dell’anticodone non legato in modo stabile facilita il distacco del tRNA dal filamento da tradurre.
Il sito P del ribosoma consente questo appaiamento impreciso, così come vale per la stessa aminoacil-tRNA sintetasi. L’enzima infatti può riconoscere l’RNA transfer corretto anche con sole due basi ben appaiate. Avere una minore specificità abbassa i costi energetici a livello cellulare, perché è sufficiente produrre un numero più basso di tRNA.
A livello del terzo nucleotide per esempio è consentito un appaiamento anche tra una guanina (G) e un uracile (U) al posto della citosina. Si tratta in fondo sempre di una purina che si unisce a una pirimidina quindi la lunghezza del legame non risulta compromessa. Non sarebbe possibile invece unire G e A, poiché sono entrambe basi puriniche.